制限 酵素 地図 299450
55.制限酵素と連結酵素 56.Green Fluorescent Protein (GFP) とは 57.プラスミドpGLOの構造 58. アンピシリン(Ampicillin)とβラクタマゼ(β lactamase) 59.pGLOプラスミド導入大腸菌におけるタンパク質の発現 510.アラビノースオペロンと遺伝子発現調節 511.プロモータ配列と遺伝子発現 512.大腸MIKENORA RESearch® Ver 40 < Manual > 1 1.はじめに MIKENORA RESearch® は、テキストデータの塩基配列から制限酵素地図の表示ができ、各酵素名、 切断部位、切断数から目的に合った制限酵素検索を支援するソフトウェアです。Sv40 dnaの 制限分解酵素による切断地図(図1) dna分 子の制限分解酵素による分析の第一段階は, 酵素によって切断されたdnaフ ラグメントの配列をき めることであるdnaフ ラグメントは,そ の大きさに 従ってアガロースあるいはポリアクリルアミドのゲル電 気泳動法で分離されるフ ラグメントの

83 制限酵素 Lecture
制限 酵素 地図
制限 酵素 地図-① ゲノムDNAを染色体ごとに分けた後,制限酵素を用いて10万 ~万塩基対の断片に切断 ② 断片をBAC(細菌人工染色体)にクローニング後,BACクロー ンのBACライブラリを作成 ③ 物理地図を用いて,各BACクローンのゲノム(染色体)上での 位置を決定 ④ BACクローンの配列を決定するた制限酵素切断地図の作成 NCBI Genome Workbench 無償 Mac, Win対応 ApE Mac, Win対応 ApE(A plasmid Editor)を利用してプラスミドを設計する Serial Cloner Mac, Win対応 DNA Dynamo Mac, Win対応(有償 $249, a licence for one PC;



Pmw119 Dna
• pUC19の情報を取得し、制限酵素地図のイメー ジ(linear)を作成、画像ファイルを保存する。 • pUC18とpUC19の制限酵素地図をパワーポイ ントのスライドに並べてに貼り付け、スライドを作 成して下さい。 • PDFファイルに変換し、メールに添付する。 • 件名は「講義4課題1」とする。DNA上に各種制限酵素の切断個所を図示したものを制限酵素地図 restriction mapといい,遺伝子の関連性の検討 (たとえば親子鑑定) や遺伝子の解析に用いられる。制限酵素の名称は細菌の属名の最初の1文字,種名の最初の2文字の次に株名やローマ数字をつける。(2)サブクローニングの制限酵素地図を作成する。使用したベクターの制限酵素地図により、どのような手順でベクター及びインサートを調製し、ライゲーシ ョンを行ったのかを理解する。また、制限酵素(EcoR I、Pvu II)による切断について、制限酵素切断地図(物理地図)を作成しレポートに記せ(実習書p19の質問16)。
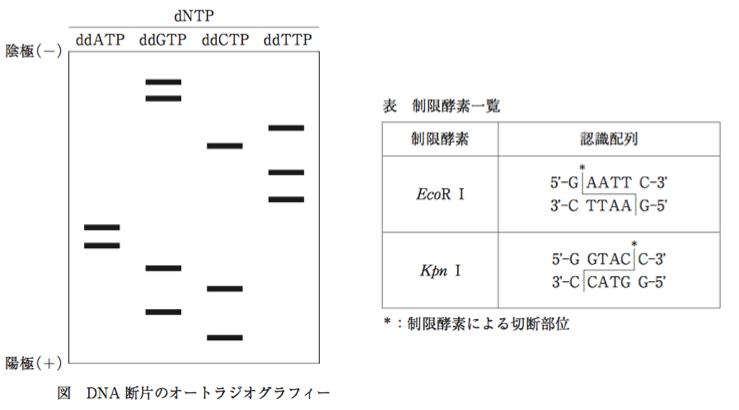
制限酵素でDNAを過剰消化した後、T4 DNA Ligaseで再ライゲーションする割合、それにリカットできる割合をアガロース電気泳動で決定。 ・ 非特異的DNase活性(16時間) DNA基質を含む反応中において、非特異的DNAの分解を試験。16時間のインキュベーションの後、アガロースゲル電気泳動簡単に言うと,制限酵素処理用の 10 倍濃度反応液が NEBuffer 1~4 の 4 種類 (プラス 特別な酵素専用の反応液がいくつか)。表では,それぞれの NEBuffer で特定の酵素を 反応させたときに,最適な反応液での活性と比べて何パーセントの活性を発揮するかを 示した表です。二つ以上の酵素の最適という風に切断します。 また例えば XhoIという制限酵素は 5'CTCGAG3'という配列を 認識して C /TCGAGという風に切断します(図 1)。 図 1に示した例は,切断された DNA の末端において 5'末端の方が 4 塩基突出した形になっています。 このような末端を付着末端 (cohesive end)あるいは 5' 突出末端(5'protruding end)といいます。 酵素の種類によって は,逆に 3
教養と学問、サイエンス > 生物、動物、植物 制限酵素地図の書き方を教えてください。 制限酵素地図の概念というかルールがいまいちわからないの ルールがいまいちわからないのですが、これは例えば8分割した横軸にABが332なので横軸を3、2、3のように分割できるようにAを3と5にBを8に書くものだと思っていたのですが、この解釈だとCが入っている場合の時 制限酵素地図を書く問題です。大学で練習問題が出されたのですが何がなんだが意味が分かりません。 10KbpのpDNAを3種類の制限酵素で切断した結果、以下のような切断パターンになった。制限酵素地図を作製しなさい。 ①酵素A、10Kbpにバンド1本 ②酵素B、10Kbpにバンド1本 ③酵素C、10Kbpにバンド1本 ④酵素AB、2Kbpと8Kbpにバンド ⑤酵素AC、4Kbpと6Kbpにバンド ⑥酵素 SnapGene Viewer Free software for plasmid mapping, primer design, and restriction site analysis Make plasmid maps automatically, browse chromosomes, view and edit sequence traces, and share annotated DNA sequences with colleagues or customers wwwsnapgenecom



Http Www Akita C Ed Jp Center Action Plan H24pdf High 80 Pdf



Egfp と プラスミド の配列
モチーフ検索 プラスミド地図作成 核酸配列自動結合 波形のサポート;PBR322 DNA(4361 bp)の制限酵素切断地図 数字は、 Eco R I認識部位の中央を0として、その位置から制限酵素の最初の認識塩基までの数を表した。 円の内側にはpBR322を1ヵ所切断する制限酵素を列挙した。 外側には2ヵ所と3ヵ所切断する酵素を列挙し、( )内はその制限酵素の切断数を示した。 GenBankへの登録 Accession No:J 制限酵素切断地図はPDFデータをダウンロード従来の定義では、制限酵素1ユニットは最適条件下で規定の基質1 μg(例:プラスミドpUC19)を1時間で切断し50 μLにします。 ユニット定義により測定形式が提供されますが、同量の制限酵素存在下での様々なDNA基質は、使用したDNA基質および基質タイプ中の認識シーケンス頻度に応じて、異なる最適要件を持っている場合があることに留意してください。 実際には、DNA



制限酵素によるプラスミドの切断



1
制限酵素地図・問題に落とし込むまで 今回は珍しく(? )医学or生物学関連のことについて書きます。 生化学の実習で制限酵素地図というものを作りました。 題材としてはかなり典型的なものだと思います。 制限酵素というのは、特定の配列の部分を制限酵素は、二本鎖 dna の特定の塩基配列を認識して結合し、切断する。切断に必要な因子や切断の仕方(位置)によって i~iii 型の 3 種類に分けられる。i 型、iii 型は切断部位が一定でないのに対し、ii 型は特定の部位を切断するため、一般に遺伝子組換え実験には ii 型酵素が用いられる。 ii制限酵素というのは、特定の配列の部分を切り取ることができるもので、制限酵素地図を書くというときは、 大体大腸菌と sv40 dnaの 制限分解酵素による切断地図(図1) dna分 子の制限分解酵素による分析の第一段階は, 酵素によって切断されたdnaフ ラグメントの配列をき めることであるdnaフ



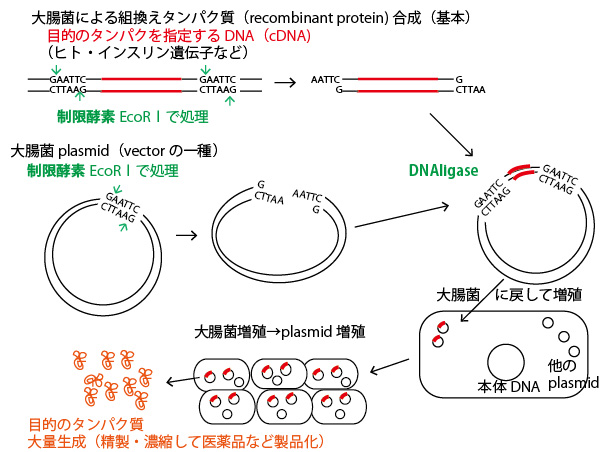
遺伝子工学の技術



生物学的安全性が向上した経口ワクチン接種用凍結乾燥形態のコレラ菌弱毒化株
Ii 型制限酵素 ii型の酵素は4~8塩基対の回文構造を認識するものが多い。400種以上知られているが、その内100種ほどが市販されている。遺伝子工学になくてはならない酵素である。 表 2のように、分解の様式から、付着末端を生じる酵素群と、平滑末端を生じる酵素群に大別される。i型やiii型と$649, a licence for 4 PCs) 無料デモ版あり 分子系統解析・ 塩基多型解析ソフトウェア MEGA おすすめ 分子進化 制限酵素地図を書く問題です。大学で練習問題が出されたのですが何がなんだが意味が分かりません。 10KbpのpDNAを3種類の制限酵素で切断した結果、以下のような切断パターンになった。制限酵素地図を作製しなさい。 ①酵素A、10Kbpにバンド1本 ②酵素B、10Kbpにバンド1本 ③酵素C、10Kbpにバンド1本 ④酵素AB、2Kbpと8Kbpにバンド ⑤酵素AC、4Kbpと6K




制限酵素地図の意味 用法を知る Astamuse



走れdna 遺伝子の部屋 Ns遺伝子研究室
制限酵素地図を作成する。(グラフィック出力) MAPSELECT オプションで設定した制限酵素のリスト・ファイルを作る。 以下の起動上の option はすべての MAP関連 program (MAP, MAPSORT, MAPPLOT, MAPSELECT)で共通である。 % map onc sequence上を一ケ所だけ切る enzyme のみを使用する場合 % map six 6base 認識の enzyme各プラスミドDNAの 制限酵素地図を示す。ただしpSC101(=)中 には HaeII部 位が10か 所以上あるが,図 には自律増殖能を示すHaeII断 片(〓) のみを示す。pKY135(―)はpBR322中Ampr遺 伝子と複製開始領 域を含む2個 のHaeII断 片から成る "ミニpBR322" である。pKMY2511b08 高等学校における分子生物学実験の実践 プラスミドdnaの制限酵素地図の作製(一般研究発表(口頭発表),日本理科教育学会第59回全国大会) 著者 遠藤,金吾他 出版者 日本理科教育学会 出版年月日 掲載雑誌名 日本理科教育学会全国大会発表論文集




制限酵素地図の作成 3 2kbのプラスミドの制限酵素部位aおよびeに 制 Okwave




解き方分かる方お願いします Clear
PUC19 DNA(2686 bp)の制限酵素切断地図 数字は、TCGCGCGTTTの最初のTを1として、その位置から制限酵素の最初の認識塩基までの数を表した。 円の内側にはpUC19を1ヶ所切断する制限酵素を列挙した。 外側にはクローニングサイトおよび2ヶ所と3ヶ所切断する酵素を列挙し、( )内はその制限酵素の切断数を示した。 GenBankへの登録 Accession NoL 制限酵素切断地図はPDFプラスミド地図を手軽に作成するプログラムです。制限酵素の位置や遺伝因子の部位を入力すると自動的に地図を描きます。地図のデザイン変更も容易に行えます。 html を使った書式設定で、文字列の一部を強調したり色づけできます。任意のウェブページへのリンカーも作れます。 原点の制限酵素を使ってDNA分子の地図をつくりましょう。 やぁ! フィル・シャープは 最初に制限酵素を使ってDNA 分子を"マッピング"したひとりでした。15,000塩基対 (15 kb)の長さの細菌DNAがここにあるとします。 この15 kbのDNAを3つの制限酵素、BamHI、EcoRI、HindIIIを使ってマッピングしましょう。15 kbのDNAを3つの酵素のそれぞれで切断できるように3つの反応を行います。それ




書き方を教えてください Clear



薬剤師国家試験 第103回 問115 過去問解説 E Rec わかりやすい解説動画
シーケンスアセンブラー 対応機種 Microsoft Windows 7 / Vista / XP が動作する機種 OS 今回はCLC Sequence Viewerの制限酵素切断部位検索機能を用いてヒトFerrochelataseの制限酵素切断地図を作成し、画像として外部出力する方法を紹介します。今回使用した塩基配列情報は dbgetから見ることが出来ます。 関連動画 この動画のタグ 環境設定;



単離dnaの制限酵素地図の作成の問題です 助けてくださる方がいらっしゃいま Yahoo 知恵袋



Http Www Akita C Ed Jp Center Action Plan H24pdf High 80 Pdf




制限酵素地図の意味 用法を知る Astamuse




83 制限酵素 Lecture



Genetyx Ver 10 Genetyx Corporation




1994 号 シャトルベクター Astamuse



大学で制限酵素地図作成の課題が出されました どこから手をつければよいか分かり Yahoo 知恵袋




プラスミド地図作成 リニア サーキュラ Genetyx Ver 8 機能説明




Puc18 Dna



遺伝子工学の技術



bkウイルスエンハンサーを含有する真核生物性発現ベクターを用いる方法



1



bkウイルスエンハンサーを含有する真核生物性発現ベクターを用いる方法




高校生物 遺伝子組換え 映像授業のtry It トライイット



In Silico Biology Com Imc I01k 制限酵素地図を描く メインフィーチャーマップ上の制限酵素レーン




この問題の 2 がわからないです Clear




制限酵素認識部位解析および制限酵素地図作成 Genetyx Ver 8 機能説明




基礎生物工学演習 の過去問集



制限酵素地図の書き方を教えてください 制限酵素地図の概念というか Yahoo 知恵袋



Www Bio Rad Com Webroot Web Pdf Lsr Japan Japanese Literature Kit 5 M4123 1303e Pdf



ある環状プラスミドを3種類の制限酵素で切断したとき 下表のような Yahoo 知恵袋




Puc119 Dna 3162 Bp の制限酵素切断地図 タカラバイオ株式会社
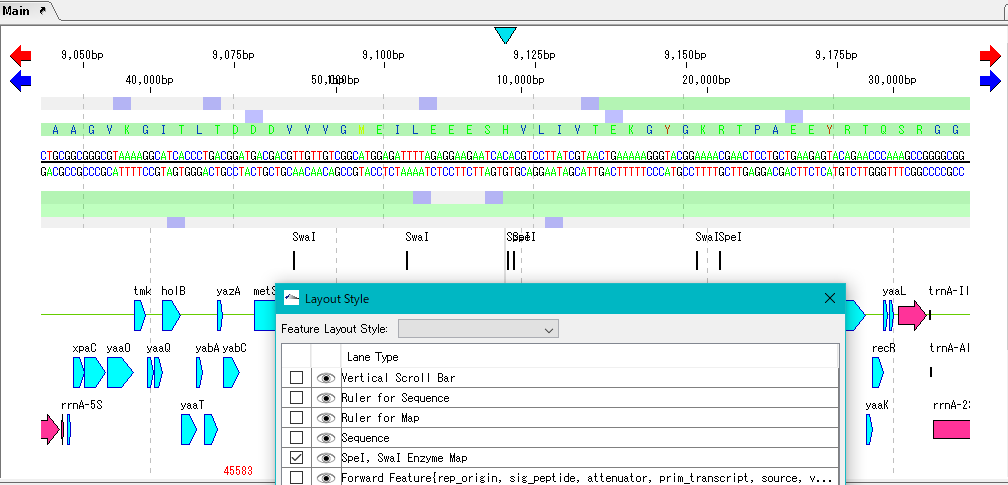



In Silico Biology Com Imc I01k 制限酵素地図を描く メインフィーチャーマップ上の制限酵素レーン



高発現ベクター バチルス属細菌 およびペプチド又は蛋白質の製造方法



遺伝子工学の技術




制限酵素地図の意味 用法を知る Astamuse



遺伝育種学研究室
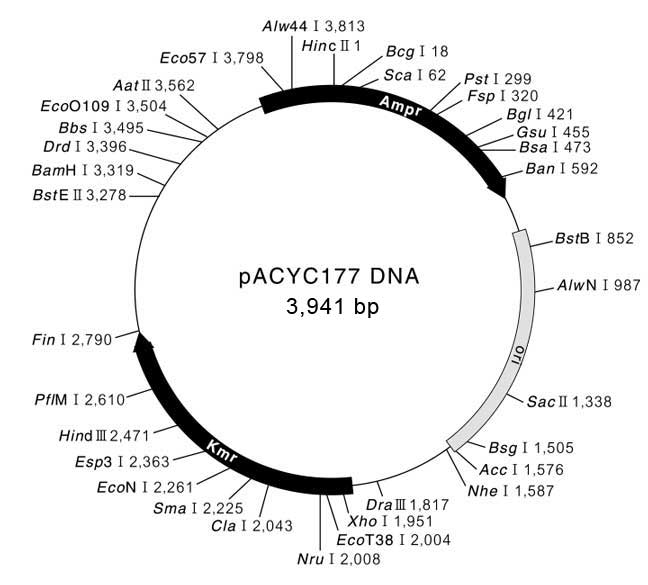



Pacyc177 Dna




新規開発の制限酵素断片長多型 Rflp マーカーを含むオオムギrflp地図 Agriknowledge



Catalog Takara Bio Co Jp Pdfs Puc119 Pdf
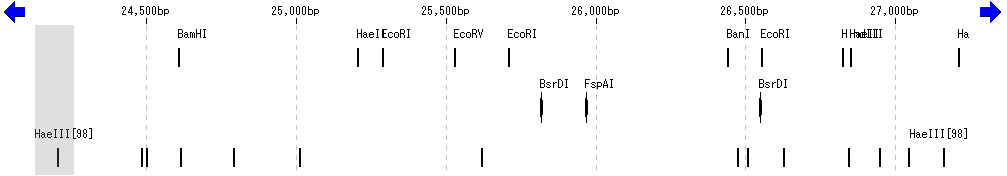



In Silico Biology Com Imc B2g 制限酵素地図レーンの機能と操作




制限酵素切断地図の意味 用法を知る Astamuse




高校生物 制限酵素切断地図 3種類の制限酵素で切断される直鎖状dnaの場合 Youtube
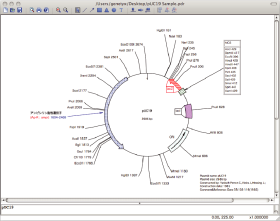



プラスミド地図作成 Genetyx Mac Ver 15 新機能 向上点



学位論文要旨詳細
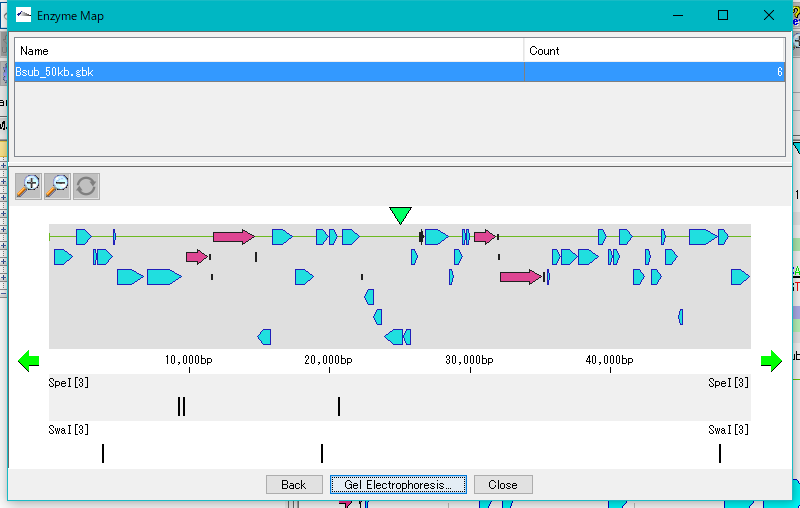



In Silico Biology Com 制限酵素地図ウィンドウ



18年大学入試センター試験 生物 第6問 遺伝子組み換え技術 選択 配点10点 問題 解答 解説 船橋市議会議員 朝倉幹晴公式サイト



Pcr 産物の制限酵素処理



Q Tbn And9gcsjc8ax U3emi Qbstnvgkx8fqy2uqgltpf42nzyiyw25b4rcka Usqp Cau



Egfp と プラスミド の配列




解き方分かる方お願いします Clear




プラスミド地図作成 環状 線状 Genetyx Ver 12 機能説明



192 10月28日 ヌクレオチド伸長反応 制限酵素 Dnaリガーゼ作用部位
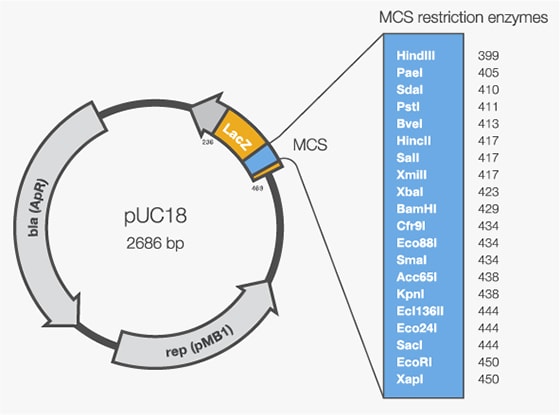



クローニングの基礎知識 Thermo Fisher Scientific Jp




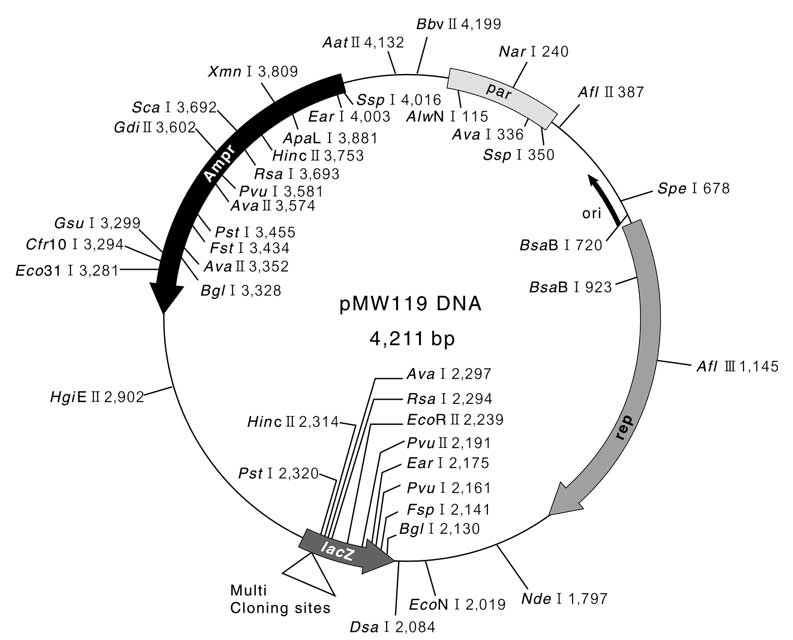
Pmw119 Dna



2
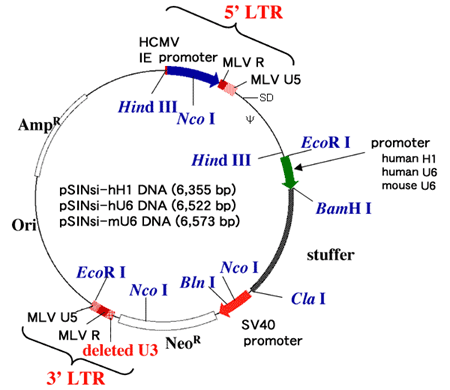



Psinsiベクターシリーズ タカラバイオ株式会社



Core Ac Uk Download Pdf Pdf
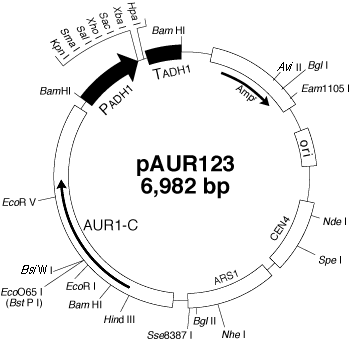



Paur123 Dna タカラバイオ株式会社



高校生物の制限酵素地図について 問題が下になります とて Yahoo 知恵袋



Http Web Tuat Ac Jp Idenshi Japanese files Seminar Folder Rikakyoin Folder H21 Folder Archives h21 H21kensyu4 5 Text Pdf
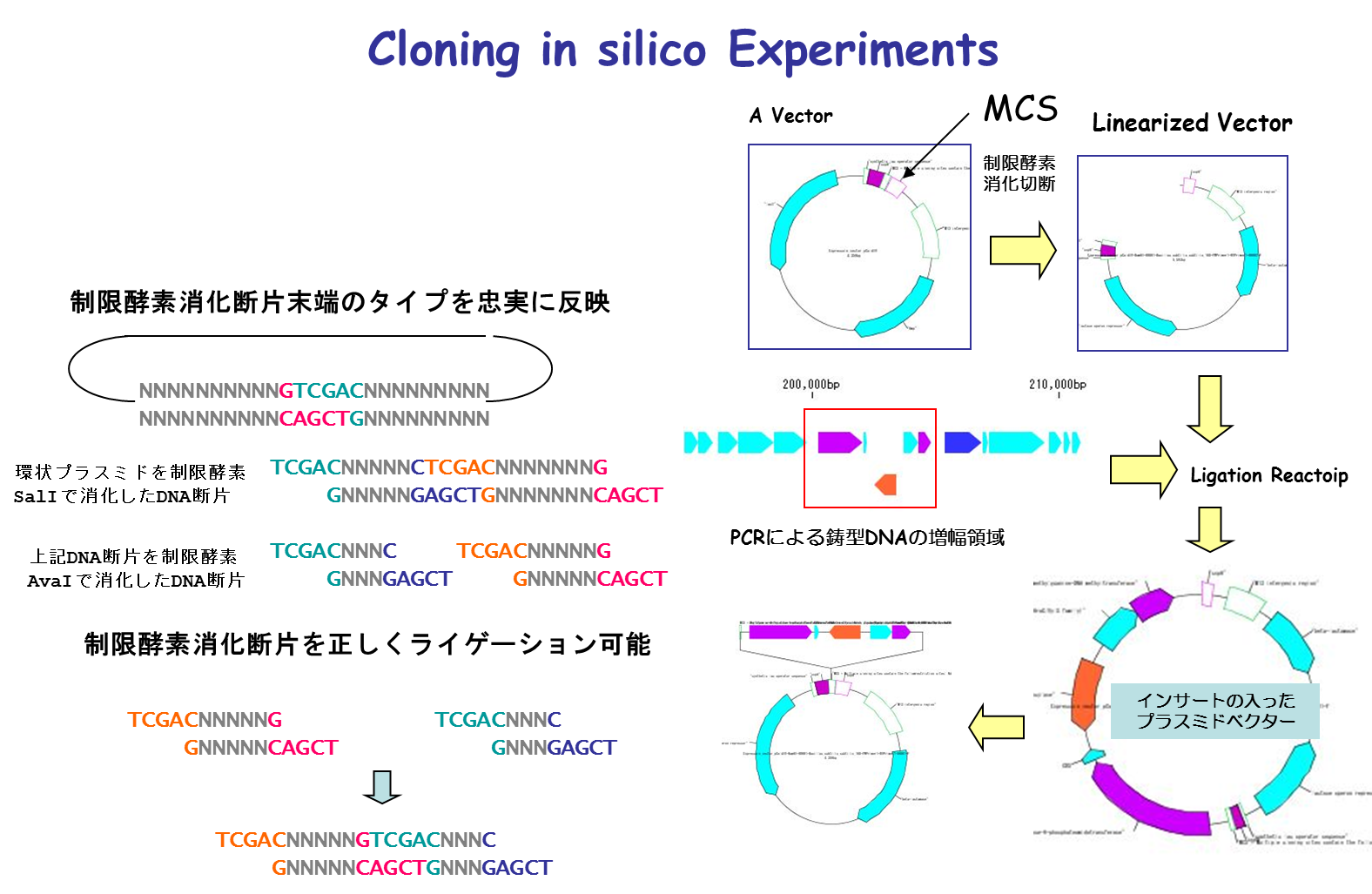



In Silico Biology Co Jp クローニング インシリコ



プラスミド の配列



制限酵素地図 に関するq A Yahoo 知恵袋



Core Ac Uk Download Pdf Pdf



クイズの解答発表 ー8 30高校オープンスクール理科 三田国際学園中学校 高等学校



Http Bunshi4 Bio Nagoya U Ac Jp Bunshi4 Class 06kyouyou 0426 Pdf



In Silico Biology Com Imc I01k 制限酵素地図を描く メインフィーチャーマップ上の制限酵素レーン




これはどうやって求めるのですか 1枚目が問題で 2枚目が答えです Clear




制限 酵素 地図 作成 ソフト



Pbr322 Dna 4361 Bp の制限酵素切断地図 タカラバイオ株式会社



遺伝子工学の技術
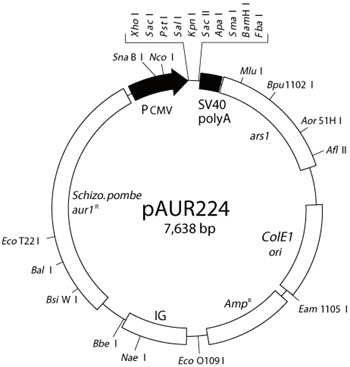



Paur224 Dna タカラバイオ株式会社




制限 酵素 地図 高校 生物




Woa1 機能改善したrnaポリメラーゼ変異体 Google Patents




1994 号 フェノールハイドロキシラーゼ遺伝子領域を含むdna断片 組換プラスミド 形質転換体およびトリクロロエチレンの分解方法 Astamuse



学位論文要旨詳細



戦いは続く 最近どうなのよ 俺 byとってぃ



Q Tbn And9gcrmnu8hnlhorejqo0ctqypdq Iubdt38wenqs4yoy8mmmb6gwoh Usqp Cau



デスクトップ クローニング ツール Gene Construction Kit ヒューリンクス




00 号 制限酵素及びその遺伝子 Astamuse



プラスミド の配列



制限酵素地図 に関するq A Yahoo 知恵袋



デスクトップ クローニング ツール Gene Construction Kit ヒューリンクス



クイズの解答発表 ー10 3高校オープンスクール理科 三田国際学園中学校 高等学校



遺伝子工学の技術



バイオテクノロジーと Genomics



走れdna 遺伝子の部屋 Ns遺伝子研究室



遺伝子工学の技術



In Silico Biology Com Imc F06f 制限酵素断片をインサートしたプラスミドマップを吹き出し表示する
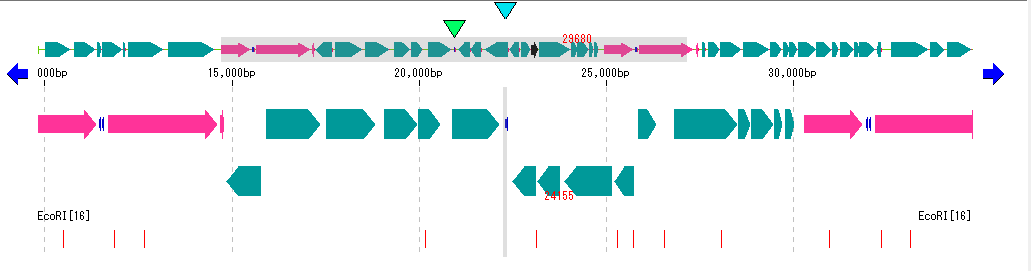



In Silico Biology Com Imc B2g 制限酵素地図レーンの機能と操作



Pbr322制限酵素地図書き方



Gair Media Gunma U Ac Jp Dspace Bitstream 16 1 24 13 Pdf
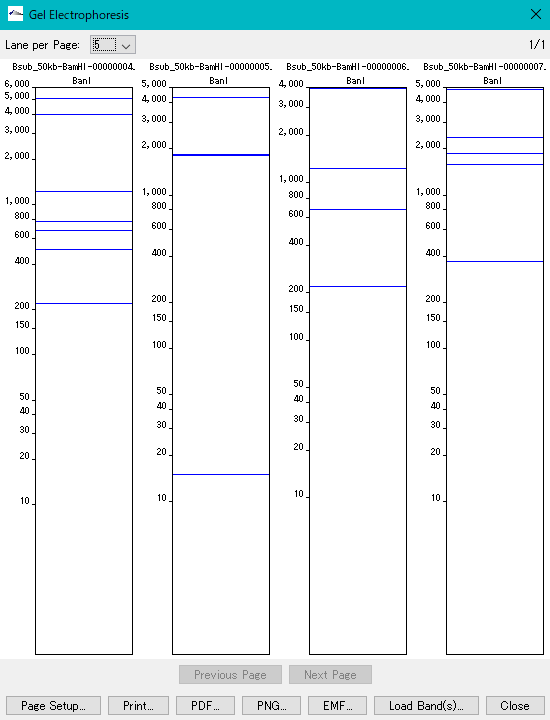



In Silico Biology Com Imc Ix01 複数のdna配列を制限酵素消化切断 ゲル電気泳動 バンド切り出し
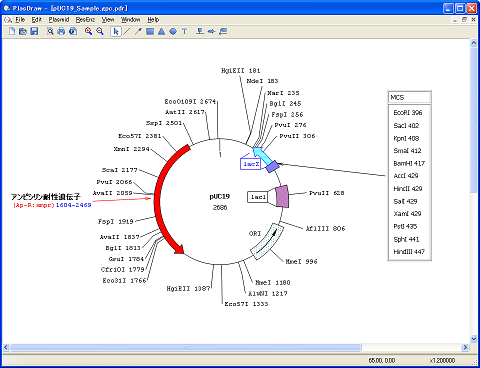



プラスミド地図作成 環状 線状 Genetyx Ver 10 機能説明
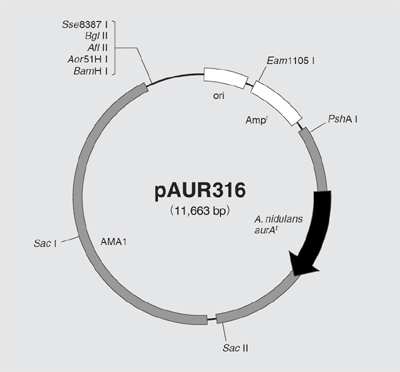



Paur316 Dna タカラバイオ株式会社
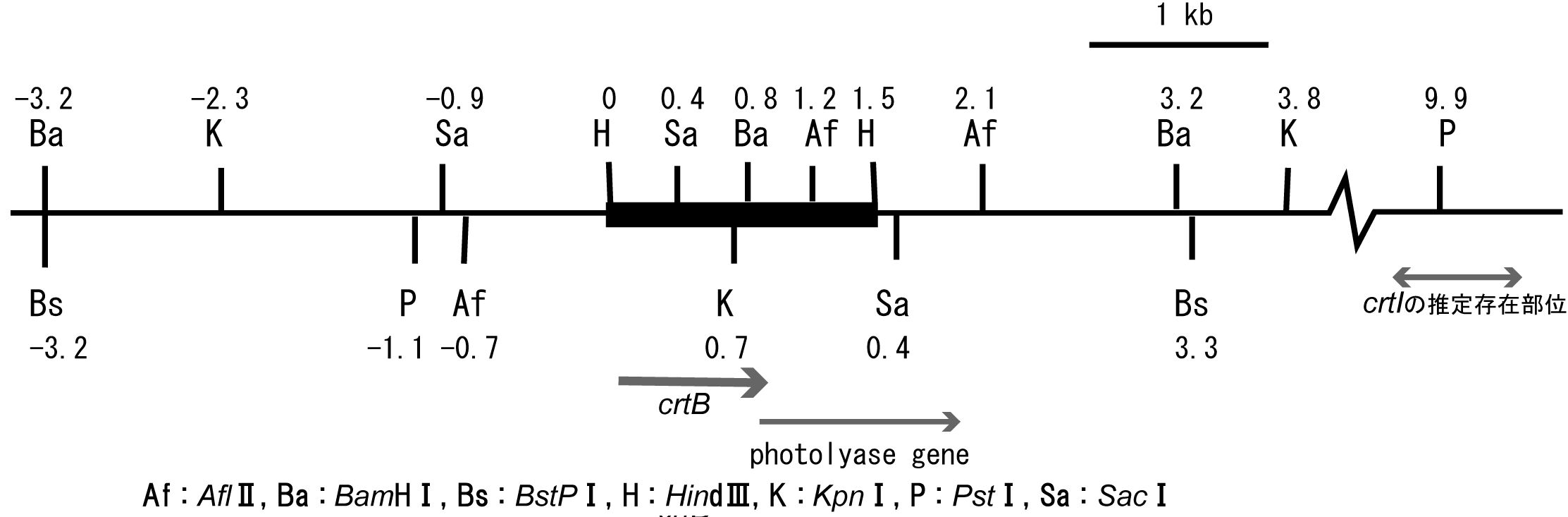



Tjb
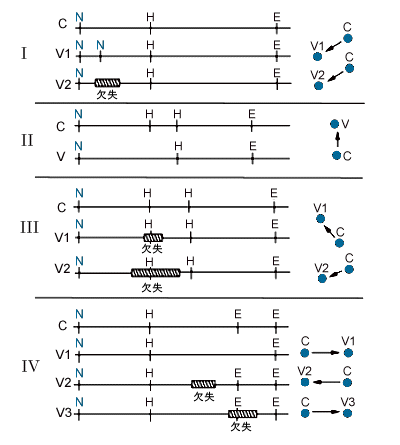



末端標識したゲノムdna断片の2次元電気泳動法 公益財団法人 放射線影響研究所 Rerf



やぶにらみ生物論83 制限酵素 渋めのダージリンはいかが



遺伝子工学の技術
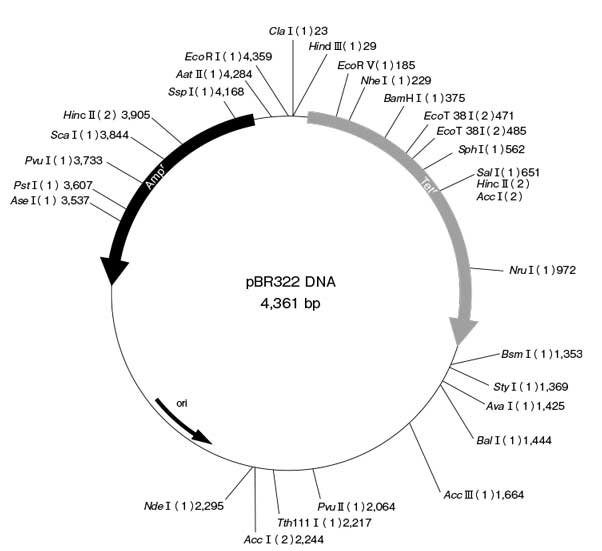



Pbr322 Dna




生物系薬学演習 第8回 中西1 Flashcards Quizlet


コメント
コメントを投稿